據中國農科院最新消息,該院作物科學研究所水稻分子設計技術與應用創新團隊,構建了一個全基因組基因功能單倍型(gcHap)數據集,全面揭示了亞洲栽培稻基因功能單倍型自然變異特征,提出亞洲栽培稻多起源(馴化)假說,并開發了適用于功能單倍型數據全基因組關聯分析和全基因組預測的軟件包——HAPS。相關研究成果新近發表于《分子植物(Molecular Plant)》。
團隊成員、中國農科院作科所研究員徐建龍介紹,目前,關于水稻種內2個亞種indica (秈)和japonica (粳)起源的假說主要有二:一是單起源假說,認為亞洲栽培稻是從單一的野生祖先起源,然后為適應不同的生態和地理環境分化為秈和粳2個亞種;二是多起源假說,認為秈和粳獨立起源于不同的野生祖先類群。而在生產上,加速育種進程、提升高產潛力和穩產性、兼顧品質改良等,是目前的迫切需求。
為加快水稻功能基因組研究與水稻分子育種的融合,該團隊于2018年牽頭完成了“3000份水稻基因組計劃”,從三個主要方面揭示了水稻核心種質的基因組多樣性:超過3200萬個高質量SNP;3萬多個水稻編碼基因上的存在-缺失變異;超過9.3萬個結構變異。然而,作為水稻種內遺傳多樣性的另一個重要方面,基因編碼區的功能單倍型(功能等位基因)在水稻種質資源中的變異特征尚不清晰。
在該項研究中,研究人員根據“日本晴”參考基因組注釋信息,基于3010份水稻核心種質基因編碼區的非同義SNP數據,構建了一個由45963個基因組成的全基因組基因功能單倍型數據集。全面揭示了亞洲栽培稻基因功能單倍型自然變異特征,提出亞洲栽培稻多起源(馴化)假說,評估了現代育種對全基因組功能單倍型多樣性的影響,對已克隆基因的有利功能單倍型進行了深入挖掘。發現基于功能單倍型數據在全基因組關聯分析較SNP數據有更大的功效,在多數性狀上具有更高的預測力,并開發了適用于功能單倍型數據全基因組關聯分析和全基因組預測的軟件包“HAPS”,為今后水稻基因的基礎研究和復雜性狀有利等位基因發掘提供極大便利,全面助力水稻分子設計育種。同時也對其他作物群體基因組、功能基因組和設計育種研究具有借鑒意義。
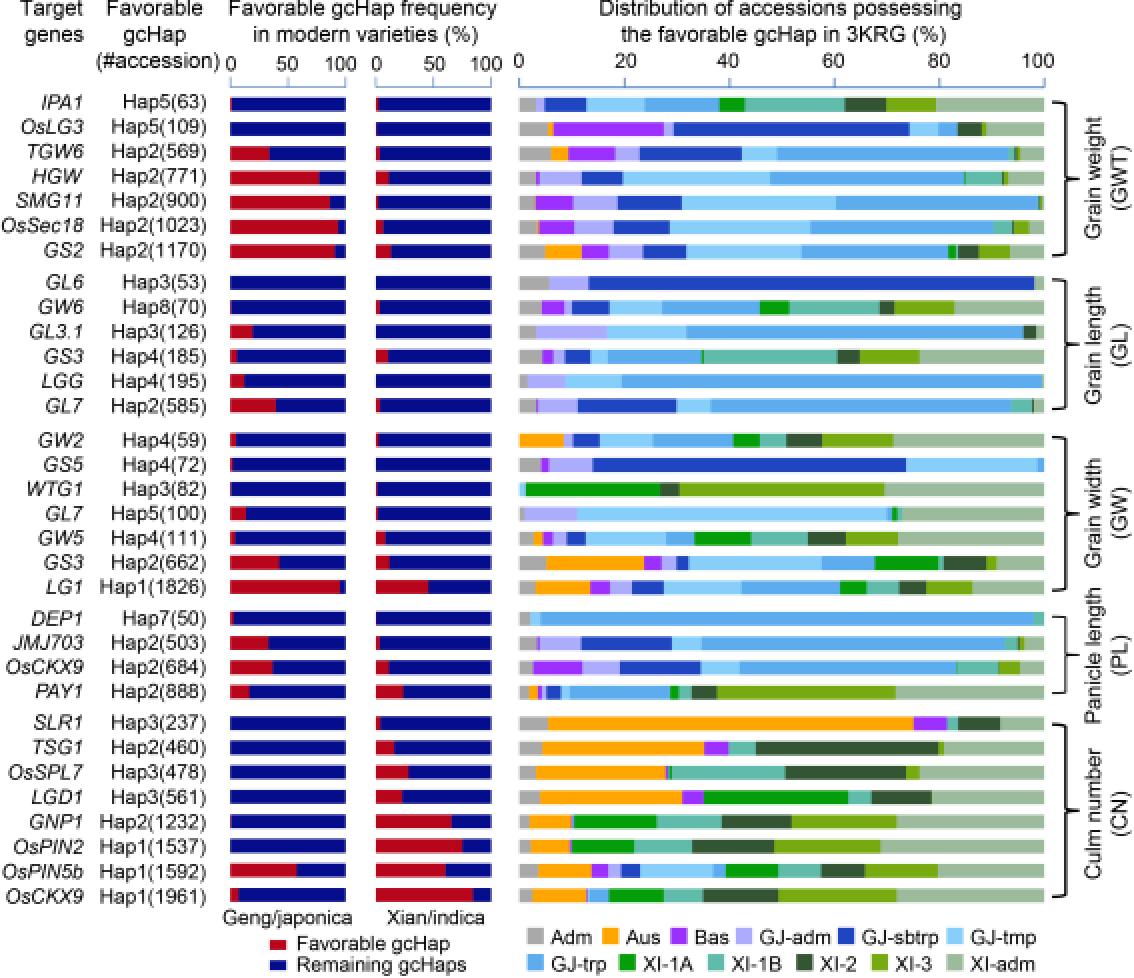
產量相關基因“有利”gcHap在秈粳育成品種中的頻率及其在種質資源中的分布。