十年前,Illumina基因組測序技術進入市場時,前所未有的龐大數據量淘汰了較早開發的測序分析工具。
歷史總是重演。如今,第三代測序技術已經達到低成本群體測序規模的臨界點。
英國時間12月9日,《自然—方法學》在線發表了第一個能夠跟上基因組測序產生速度的組裝算法。
這篇論文只有兩位作者,他們是中國農業科學院農業基因組研究所阮玨博士與美國哈佛大學醫學院李恒博士。這個新的第三代測序數據組裝算法被他們稱為Wtdbg。
三代測序的尷尬
20年前,破譯人類遺傳密碼還是極具挑戰的大科學工程,當時的人類基因組測序計劃與曼哈頓原子彈計劃、阿波羅計劃并稱為三大科學計劃。
如今,完成一個人的全基因組測序已經是普通實驗室甚至家庭都可以負擔起費用的“平常”事情。用第三代測序技術完成個體全基因組測序僅需一天時間,費用也已經低于5萬元。
2011年,PacBio公司正式宣布三代單分子測序開始商業化。相比于二代測序每個序列的幾百堿基對測序讀長,三代測序的平均讀長達到了幾萬堿基對,最長可以達到數百萬堿基對。
西北工業大學生態環境學院教授邱強告訴《中國科學報》,這一技術出現時,科研人員期待利用它填補基因組序列中高重復高雜合的區域,挑戰高難度的基因組。然而,人們迅速發現,這一新技術的普及和應用遇到了很大的困難。
“這主要由兩個原因所導致。第一,三代測序的成本在初期要遠高于二代測序;第二,由于三代測序錯誤率較高,此前用于第二代基因組測序的組裝方法紛紛失效,缺乏有效率的組裝工具,特別是PacBio官方推出的falcon方法,消耗資源極多。”邱強介紹,數年后,Ont公司推出納米孔測序技術,市場競爭逐漸拉低了第三代測序的成本。而在基因組組裝方面,盡管已經出現了canu、marvel等多個組裝軟件,“但組裝仍然是一個十分費時費力的過程,一個哺乳動物基因組的組裝時間要以數周來計算”。
以人類基因組組裝為例,在2014年需要消耗50萬個CPU小時,只能在超大計算機集群上進行。“這種情況下,同時對大量個體進行組裝分析是難以想象的。”但現實是,“以全基因組組裝方式對群體進行測序分析已經成為生物醫學研究的趨勢”,阮玨說。
首次:數據分析比數據產生更快
“wtdbg和即將推出的工具可能會從根本上改變當前測序數據分析的實踐。”阮玨在接受《中國科學報》采訪時說。
此前,“數據產出速度遠高于數據分析速度。”因此,近年來,生物信息學領域的科學家群體致力于改變這種尷尬狀況,不斷開發出更高效的組裝分析算法。
例如,繼falcon、canu等算法之后,2019年4月,美國加利福尼亞大學圣地亞哥分校NIH計算質譜中心主任Pavel A. Pevzner在《自然—生物技術》上發表了Flye算法,其速度遠高于falcon、canu。
而阮玨和李恒正式發表的第三代測序數據組裝算法wtdbg,比之Flye算法,分析速度提升了5倍,也首次讓數據分析時間少于數據產出時間。
西北工業大學生態環境學院的科學家已經用wtdbg組裝了十多個哺乳動物基因組。西北工業大學教授陳壘在接受《中國科學報》采訪時說:“我們用過falcon和canu等組裝方法,相比較而言,wtdbg組裝運算時間最快,占用資源少,能節省大量時間。組裝出的基因組連續性很高,組裝質量均符合現在主流的基因組評估。”特別是,對超大型基因組的組裝,wtdbg應該是目前為數不多的可以高效使用的組裝軟件。
“對于人類基因組數據,wtdbg比已發布的工具快幾十倍,同時實現了相當的連續性和準確性。它代表了算法上的重大進步,并為將來群體規模的組裝分析鋪平道路”阮玨說。
模糊布魯因圖問世
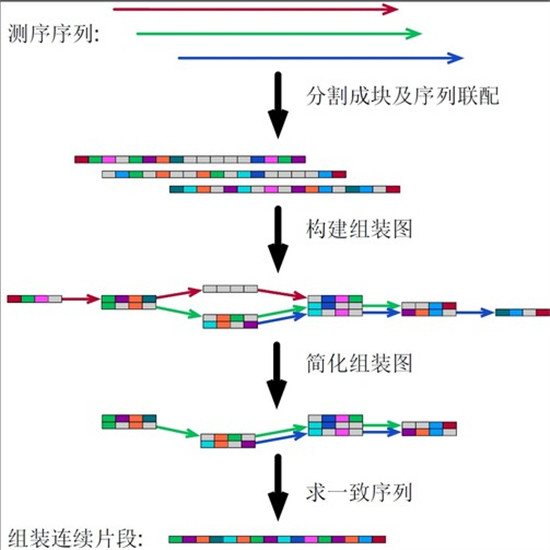
上世紀90年代,Pavel A. Pevzner將德布魯因圖(de Bruijn Graph)引入了基因組組裝領域。阮玨介紹,由于第二代測序錯誤率低,大部分短串(k-mer)是正確的,相同的短串間可以利用德布魯因圖的原理合并起來構成組裝圖。
但三代測序數據的錯誤率非常高,如果還是使用短串k-mer的話,大部分短串帶有測序錯誤,不可以合并起來。因此,德布魯因圖從未成功應用在第三代測序數據。
突破性的方法基于突破性的理論基礎。
2013年開始,阮玨和李恒著手解決三代測序組裝的問題,分別開發的SMARTdenovo和Miniasm在領域內均有較好的表現。隨后在德布魯因圖基礎上,設計出一個新的組裝圖理論——模糊布魯因圖(Fuzzy Bruijn Graph)。他們重新定義了“短串”,將測序數據切分為固定長度的新型短串k-bin,k-bin比k-mer的長度更長,“新設計的模糊布魯因圖能夠容忍高噪音數據,并隨后對生成組裝圖與恢復基因組序列做了大量相應的重構,使其兼具高效率和高容錯的優點”阮玨說。
“一般軟件組裝第三代測序數據的思路是,先對測序數據進行比對糾錯,再進行基因組序列的構建。”邱強說,wtdbg則直接進行基因組組裝,避免了需要提前糾錯的耗時步驟,直接得到一個相對可靠的組裝結果。
“組裝費時費力這一問題的真正改善,正是由阮玨和李恒所研發的wtdbg算法開始”邱強說。在他們的課題組中,wtdbg算法得到了廣泛使用,極大提高工作效率。不僅如此,他們還與阮玨進行了深入溝通,對超大基因組組裝進行了優化,“我們得以獲取基因組大小40G左右的高質量基因組序列”。
公眾參與下的技術改進
2016年,為了讓基因組測序領域可以及時使用新技術,阮玨和李恒將wtdbg研究成果對所有人免費開放使用。
3年來,wtdbg不僅被幾十篇學術論文引用,還被國內多家基因組測序分析公司作為主要組裝分析工具,并且在2019年世界大學生超算競賽中做為性能測試賽題。
“我們通過郵件、GitHub網站等方式收到大量反饋,這些反饋不僅幫助我們修訂算法軟件中的漏洞,還給我們帶來新的想法和思路。換個角度來講,現在發表的論文已經經歷了3年多的‘公眾審稿’,感謝多年來參與和關注wtdbg開發的同行。”阮玨說。
邱強認為,wtdbg算法不僅相對于更早的falcon、canu等算法具有效率和準確性的優勢,相比此后出現的flye等組裝算法也具有更好的可靠性。“這一研究成果代表我國在基因組算法領域具有不輸于國際甚至引領國際的實力,也代表了我國科技發展的軟實力”。
現在,科學家們可以使用全基因組組裝的方式,對大群體開展研究了。
相關論文信息:https://doi.org/10.1038/s41592-019-0669-3