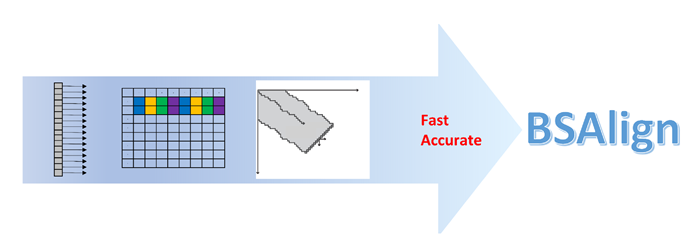
近日,中國農業科學院深圳農業基因組研究所農業基因組學技術研發與應用創新團隊提出DNA比對新算法,并研發出DNA比對軟件“BSAlign”。相比同類并行算法,新算法能夠更快生成最優比對結果,且準確度更高。相關研究成果發表在《基因組蛋白質組與生物信息學報(Genomics, Proteomics & Bioinformatics)》上。
經典的動態規劃算法,如史密斯-沃特曼算法和尼德曼-翁施算法,常用于處理序列比對,但由于其時間復雜度呈二次函數式增長,當序列長度增加時,算法的處理時間也隨之變長,導致其在處理大規模序列比對時效率低下,嚴重阻礙了其應用。
為此,研究人員提出了一種新的DNA序列對比算法——條紋移動法,該算法能夠在帶寬環境下實現高效運算。經測試,與現有并行算法相比,BSAlign算法的速度提升了2倍,在長序列比對方面,其效率較基于編輯距離的比對算法提高了1.5到4倍,在帶寬環境下實現了高效運算。此外,研究人員還進一步提出了主動F循環法,解決了條紋數據在長插入或刪除情況下的多次查詢問題,進一步提高了處理速度和準確性。
該研究得到了國家重點研發計劃、國家自然科學基金、中國農業科學院科技創新工程等項目的資助。(通訊員 馬昕怡)
原文鏈接:https://academic.oup.com/gpb/advance-article/doi/10.1093/gpbjnl/qzae025/7628627